来自 Peng Zhao | January 31, 2020
疫情肆虐,却逢春节假期,作为一个普通人,连续享受了几天葛优躺家里蹲之后,寂寞得想找点事情做。与其冒着风险转发谣言被举报,还不如做点稳妥并且对自己有好处的事情,比如说,学一样语言。可是,又放不下对疫情的关注,无法全心投入到语言学习里。怎么办?
一个主意跳进了我的脑瓜:用 R 语言来处理疫情数据!学习编程和关注疫情两不误!
这个系列的文章,面向的是 R 语言初学者,从入门开始,以 2020 年新型冠状肺炎病毒的数据为例,一起学习如何用 R 语言进行数据处理、作图、生成可重复性报告。
本文可以和 2018 年我出版的《学R:零基础学习 R 语言》一书配合,达到入门和提高的目的。为了节省篇幅,原谅我会在本文多次引用这本书。没办法,第一次印多了,库存很多本还没消化呢。当然,你可以无视,换其他适合自己的参考资料。
动笔的时候,我不知道未来会写些什么,只是想转移一下对疫情过度的注意力。我既不懂流行病学,又不懂病毒,目前只能做一点粗浅的领路抛砖工作。欢迎感兴趣的朋友加入这个话题的写作,共同学习,说不定最后能结集成一本书。
现在,让我们开始一段有意义的征程。
准备工作
开始之前,需要有个前提条件:你得有一台电脑。操作系统可以是 Windows、macOS或者 Linux,都行,但不可以是安卓。
其次,这台电脑得能连上网。
具备这两个条件的话,一切就简单了:
- 安装 R。这是必须的。在 CRAN 上选择适合你操作系统的安装包来安装。
- 安装 RStudio。这不是必须的,然而我极力推荐。它让你在使用 R 语言的时候爽到爆。在 RStudio 的官网下载安装即可。
安装完毕后,启动 RStudio,就进入了 R 语言的环境。
从现在开始,我们要在 RStudio 里执行 R 代码。上述步骤和代码 执行方法详见《学R》第一章(点击此处免费下载)。如果你是 Windows 用户,为了避免本应出现中文的地方出现乱码,先执行下面这个代码:
Sys.setlocale('LC_CTYPE', 'Chinese')## [1] "Chinese (Simplified)_China.936"安装扩展包
顾名思义,扩展包是 R 语言的扩展功能(详见《学R》第九章),令 R 有能力完成原先很难完成的工作,如虎添翼。
我们先安装本文用得着的几个扩展包:
首先是 “httr”包,它可以让 R 轻松获取网络数据:
install.packages("httr") 然后是 “jsonlite”,它可以从网络数据里摘取 json 格式的数据:
install.packages("jsonlite")安装完毕之后,需要调用他们:
require("httr")
require("jsonlite")最后是 “tidyverse”,它可以……它是全能,一言难尽,以后遇见了慢慢说。
包只需安装一次,而加载则需要每次启动 R 时都要执行。打个比方,刚安装上的 R 语言就好比刚出生的哪吒,自带本领很大,但尚未开发出来。而 RStudio 就好比哪吒学到的三头六臂,而扩展包就好比乾坤圈、混天绫、火尖枪、风火轮这些外挂,让哪吒有本事九天揽月五洋捉鳖。
外挂只需师父发一次就以后都有了,而使用时每次都要加载一下。
你准备好上天入海了吗?
作业:请自行安装和加载 “tidyverse” 包。
获取疫情地区数据
疫情数据来自“2019新型冠状病毒疫情实时爬虫”项目。感谢作者 Isaac Lin 的慷慨贡献。
这个数据集里,最重要的是“地区”数据。我们这样来读取:
base <- 'https://lab.isaaclin.cn/nCoV/api/' # api 网址
port <- 'area' #接口
get_raw <- GET(paste0(base, port)) # 获取链接
get_text <- content(get_raw, "text") # 获取数据
ncov_area <- fromJSON(get_text) #提取数据这样,疫情概述的信息就下载并储存在 ncov_overall 里了。它是个列表,里面有两个元素。我们感兴趣的数据存在名为results的表格里:
class(ncov_area) # 查看数据类型## [1] "list"length(ncov_area) # 查看数据长度## [1] 2names(ncov_area) # 查看元素名称## [1] "results" "success"这样显示出来的模样不好看。可以用 View() 函数看起来更舒服:
View(ncov_area$results)下面,我们取出这个表格的前 7 列,把它保存到本地电脑上(见《学R》第二章):
write.csv(ncov_area$results[, 1:7], 'd:/ncov_area.csv')打开d:/ncov_area.csv这个文件,不管是用 excel、openoffice 还是 notepad,都行,就看到这个数据表了。这就是目前全球各地新型冠状病毒确诊、疑似、治愈、死亡病例的数据表。
你可能会问,为什么只取出前 7 列。这是因为第 8 列的格式比较复杂,而第 9 列以后的数据不太重要。我们以后再回过头来解决这个问题,
获取疫情概况数据
与“地区”数据类似地,我们用下面的代码读取“概况数据”:
port <- 'overall' #接口
get_raw <- GET(paste0(base, port)) # 获取链接
get_text <- content(get_raw, "text") # 获取数据
ncov_overall <- fromJSON(get_text) #提取数据这样,疫情概述的信息就下载并储存在 ncov_overall 里了。
作业:仿照“地区”数据,查看“概况数据”的数据类型、数据长度、元素名称、表格内容,并用View()函数来浏览。把“地区”数据保存到名为“ncov_overall.csv”的本地文件里,并用其他软件打开浏览。
ncov_overall 表格只有一行。你会看到各列名称和内容。顾名思义,第一列 infectSource是传染源,passWay是传播途径,dailyPic 是日图。可以把日图的链接拷贝粘贴到浏览器里看一看:
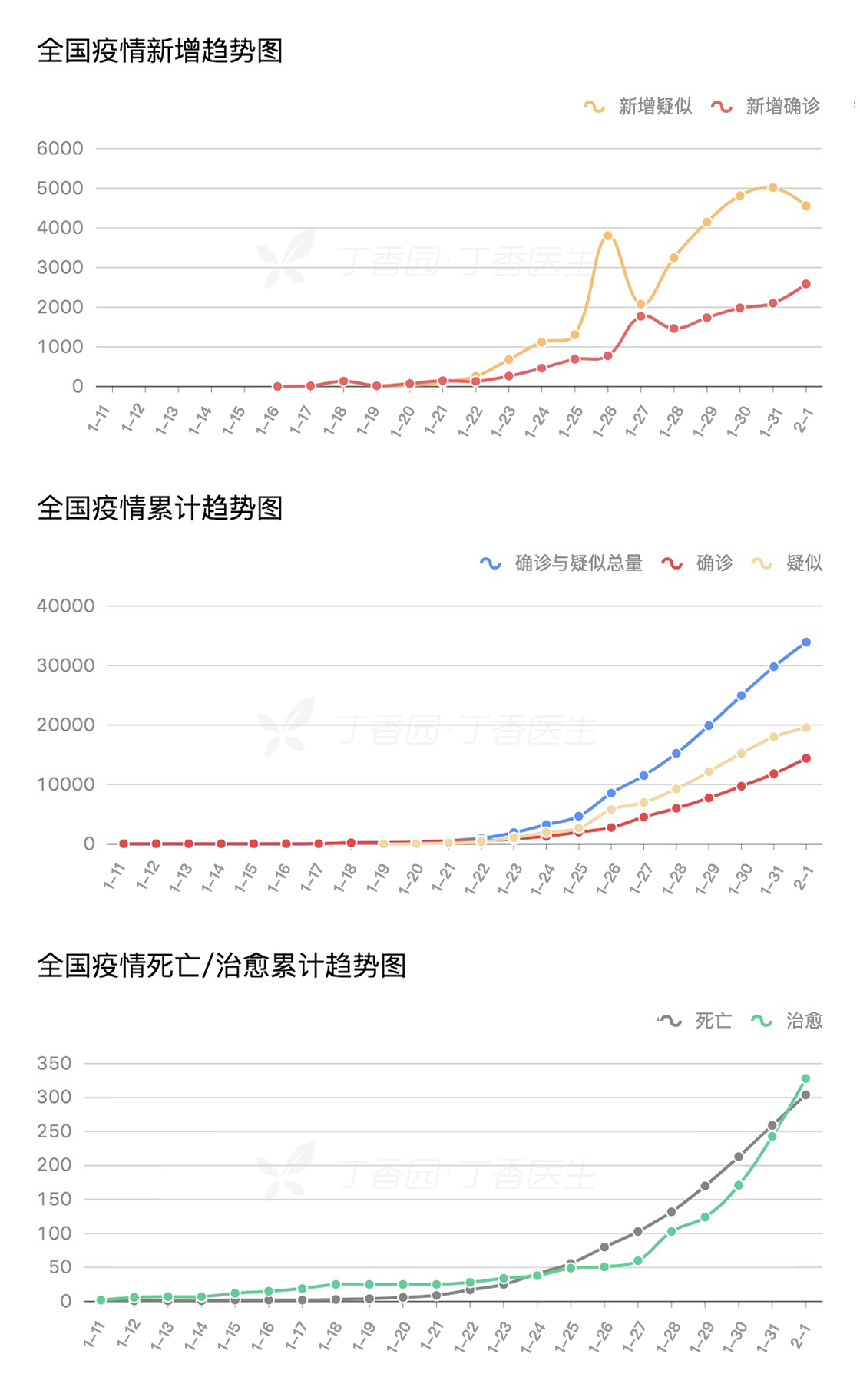
其他列的含义你猜猜看。
除了 overall 和 area,服务器还提供了 ‘provinceName’, ‘news’, ‘rumors’ 等接口,可以用来下载“省名称”、“新闻”和“谣言”数据表。
作业:请自行下载、浏览和保存 ‘provinceName’, ‘news’, ‘rumors’ 等数据。
获取全部数据
上面获取几个不同数据表的代码是类似的,只需照猫画虎就行了。把这些代码摞在一起,显得很啰嗦。如果你阅读过《学 R》第五章和第九章的话,你会想到,可以用循环或者自定义函数的方法来简化代码。
你可以试试循环语句,而我用的是自定义函数,并把这个函数写进一个包里,名叫 ncovr。有了这个包,只需一句代码,就可以下载得到上述所有数据了。
这个包发布在了 Github 上,并未发布在 CRAN 上,因此,安装方法有区别。先要安装和 remotes 包:
install.packages('remotes')
require('remotes')然后,就可以使用 ‘remotes’ 包提供的 install_github() 函数,从 Github 服务器安装 ncovr 包了:
install_github('pzhaonet/ncovr')
require("ncovr")读取所有数据:
ncov <- get_ncov()ncov 是个列表。如果查看其中的子集,例如 provinceName,只需用 $符号:
ncov$provinceName## [1] "上海市" "云南省" "俄罗斯" "内蒙古自治区"
## [5] "加拿大" "北京市" "印度" "台湾"
## [9] "吉林省" "四川省" "天津市" "宁夏回族自治区"
## [13] "安徽省" "尼泊尔" "山东省" "山西省"
## [17] "广东省" "广西壮族自治区" "待明确地区" "德国"
## [21] "意大利" "斯里兰卡" "新加坡" "新疆维吾尔自治区"
## [25] "日本" "柬埔寨" "江苏省" "江西省"
## [29] "河北省" "河南省" "法国" "泰国"
## [33] "浙江省" "海南省" "湖北省" "湖南省"
## [37] "澳大利亚" "澳门" "瑞典" "甘肃省"
## [41] "福建省" "美国" "芬兰" "英国"
## [45] "菲律宾" "蒙古" "西班牙" "西藏自治区"
## [49] "贵州省" "越南" "辽宁省" "重庆市"
## [53] "阿联酋" "陕西省" "青海省" "韩国"
## [57] "香港" "马来西亚" "黑龙江省"本节我们完成了数据的读取和保存。下一节,我们将对数据进行可视化,通俗来说就是——作图。

