来自 Peng Zhao | February 1, 2020
失眠,索性起来写第二篇。
书接上回
在上一节里,为了避免 Windows 用户遭遇中文乱码,我们将系统语言设置为中文:
Sys.setlocale('LC_CTYPE', 'Chinese')## [1] "Chinese (Simplified)_China.936"我们安装和加载了 “ncovr” 包(请重新安装一次,因为我刚刚把它升级到 0.0.2 版本了),用它来获取了疫情数据:
require(remotes)
install_github("pzhaonet/ncovr")require(ncovr)
ncov <- get_ncov()我们还安装了 “tidyverse” 包,但是并未调用它。今天,我们就要启用其中的两个成员:“dplyr”和 “magrittr”。
require(dplyr)
require(magrittr)相信大家最近这些天,对下面这张地图都很熟悉了吧:
knitr::include_graphics('https://openr.pzhao.org/img/map-ncov-netease.png')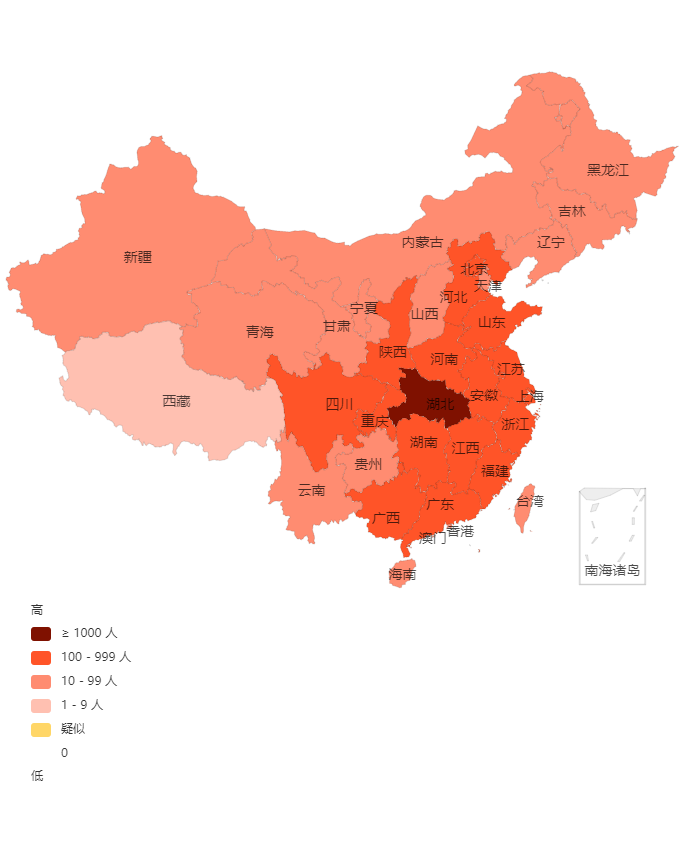
本节将以 ncov$area 数据集为基础,绘制这张地图。
准备工作
绘制地图有很多种方法(详见《学 R》第十一章:“地图”)。这里,我们选择最酷的一种,那就是交互地图,支持缩放和弹出信息的那一种。绘图所用的包是 “leaflet” 和 “leafletCN”,在上面重装 “ncovr” 包时已经自动安装了,现在只需加载一下:
require(leaflet)
require(leafletCN)准备工作完成。
试画地图
先浏览一下数据:
View(ncov$area)在 `provinceName 一列里,可以发现有个“待明确地区”,这显然不是省名称,我们先把这条记录删掉:
ncov$area <- ncov$area[ncov$area$provinceName != "待明确地区",]可能会出乎你的意料,这最酷的偏偏画起来是最简单的。只需一条指令,就可以绘制全国新型冠状病毒的确诊病例地图:
# 试画地图
geojsonMap(dat = ncov$area,
mapName = "china",
palette="Reds",
namevar = ~provinceName,
valuevar = ~confirmedCount,
legendTitle = "确诊人数")点击左上角的缩放键,就可以 zoom in/out 了。如果是触屏,可以用两个手指放大缩小。点击地图上任一位置,就会弹出对应信息(当然,如果你是在微信公众号读到了本文,是无法进行交互的。请点击文末左下角的“阅读原文”,移步网页版)。
酷吧?!
改进地图
酷是酷,这幅地图有两个问题:
- 代表确诊病例颜色的深浅是线性的。湖北省颜色最深,而其他省之间的差异不太明显。这是由于其他省跟湖北省差别很大而导致的。
- 点击各省,弹出的只有个省名称。要是能显示确诊病例数量就更好了。
对于问题 1,我们用 cut() 函数对确诊病例数进行分组;对于问题 2,只需用 geojsonMap() 函数的 popup 参数来设定:
# 修改后的地图
count_cut <- c(0, 9, 99, 999, Inf)
ncov$area$confirmed_level <- cut(
ncov$area$confirmedCount,
count_cut,
labels = c('< 10', '10-99', '100-999', '>999'))
geojsonMap(dat = ncov$area,
mapName = "china",
colorMethod = "factor",
palette="Reds",
namevar = ~provinceName,
valuevar = ~confirmed_level,
popup = paste(
ncov$area$provinceName,
ncov$area$confirmedCount),
legendTitle = "确诊人数")目标地图绘制完成!
作业:仿照上面的方法,绘制全国治愈病例地图和死亡病例地图。
超额完成
目标光完成是不够的;让我们挑战一下自我,除了完成,还要超额。
上面的地图里,虽然按数量级将各省分了组,但问题是分在同一组里的省份看不出区别。这该怎么办?
大学老师教过我们,当跨越两三个数量级并且要区分低值时,可以考虑对数坐标。
然而,查阅 “leafletCN” 包的帮助文档,却发现 geojsonMap() 函数并不支持用对数坐标来显示颜色。怎么办呢?
我抓破头皮,想了个办法,就是把 geojsonMap() 函数给黑了,重新写了个函数,叫做 geojsonMap_legendless(),收在了 “ncovr” 包里(0.0.2 版)。对,这是我刚写的,所以在本文开头请你重装了“ncovr” 包。现在,可以使用这个新函数,配合 “leaflet” 包的 addLegend() 函数,就可以绘制对数坐标图:
# 超额绘制地图
ncov$area$confirmed_log <- log10(ncov$area$confirmedCount)
ncov$area$confirmed_log[ncov$area$confirmedCount == 0] <- NA
geojsonMap_legendless(
dat = ncov$area,
mapName = "china",
palette = "Reds",
namevar = ~provinceName,
valuevar = ~confirmed_log,
popup = paste(
ncov$area$provinceName,
ncov$area$confirmedCount)) %>%
leaflet::addLegend(
"bottomright",
bins = 5,
pal = leaflet::colorNumeric(
palette = "Reds",
domain = ncov$area$confirmed_log
),
values = ncov$area$confirmed_log,
title = "确诊人数",
labFormat = leaflet::labelFormat(
transform = function(x) 10 ^ x),
opacity = 1)对数坐标的缺点在于 0 值取对数后成为负无穷大,难以展示。这里把 0 用灰色表示,跟缺少数据(NA)的区域颜色相同。
城市地图
最后,我们来做个更难的实验:
有个朋友前些天从国外回到了浙江省温州市,发现温州有了个新名字,叫“湖北省温州市”:疫情按严重程度排名的话,温州是湖北之外疫情最严重的一个城市。然而在全国地图上看不出来这个信息。
我也想看看我的家乡河南省,各市之间有什么差别。这就需要绘制以市为单元的地图了。
以 ncov$area 数据集为基础绘制城市地图,有 2 个难点:
- 城市疫情信息藏在
cities一列,形式是 list ,并不是绘图需要的 data.frame (详见《学R》第八章)。 - 然而,直辖市却存在
provinceName一列。这就需要提取出来,跟其他城市合并。
解决这两个难点的方法是这样的:
# 城市地图
ncov_city <- dplyr::bind_rows(ncov$area$cities) %>% dplyr::select(1:5)
ncov_area <- ncov$area[, 2:6]
names(ncov_area) <- names(ncov_city)
ncov_cities <- rbind(ncov_city, ncov_area)
cities <- regionNames(mapName = 'city')
ncov_cities <- ncov_cities[ncov_cities$cityName %in% cities, ]
ncov_cities$confirmed_log <- log10(ncov_cities$confirmedCount)
ncov_cities$confirmed_log[ncov_cities$confirmedCount == 0] <- NA
geojsonMap_legendless(
dat = as.data.frame(ncov_cities),
mapName = "city",
palette = "Reds",
namevar = ~cityName,
valuevar = ~confirmed_log,
popup = paste(ncov_cities$cityName,
ncov_cities$confirmedCount)) %>%
leaflet::addLegend(
"bottomright",
bins = 5,
pal = leaflet::colorNumeric(
palette = "Reds",
domain = ncov_cities$confirmed_log),
values = ncov_cities$confirmed_log,
title = "确诊人数",
labFormat = leaflet::labelFormat(
transform = function(x) 10 ^ x),
opacity = 1)温州的疫情,确实跟湖北省各市持平。
现在,运用地图的缩放功能,看看各省内部的信息吧。
作业:仿照上面的方法,绘制以城市为单元的确诊病例和死亡病例地图。
你发现没有:前面省区地图里,西藏和台湾明明是有确诊病例的,为何城市地图上没有显示出来?是不是我们画错了?
查看一下 ncov$area 数据集,就会发现,数据里缺少西藏和台湾的城市疫情数据!所以这个锅应该由源数据来背。在这个甩锅的年代,这不是我们绘图者的错。
懒人福音
本节里涉及的代码虽然结构简单,然而终究有点长,不适合懒人。要是想每天关注动态,可以用我打包封装在 “ncovr” 包里好的函数 plot_map()。忘了上面的代码吧,因为用下面的代码更方便:
Sys.setlocale('LC_CTYPE', 'Chinese')
require(ncovr)
require(leafletCN)
ncov <- get_ncov()
plot_map(ncov$area, filter = '待明确地区')
plot_map(ncov$area, filter = '待明确地区',
scale = 'log',
legend_title = '确诊病例',
method = 'province'
)
plot_map(ncov$area, filter = '待明确地区',
scale = 'log',
legend_title = '确诊病例',
method = 'city'
)如果查看这个函数的帮助,会发现更多的功能,例如可以改变颜色:
plot_map(ncov$area, filter = '待明确地区', color = "Greys")除了本文介绍的方法,绘制地图的方法还有很多,《学R》第十一章介绍了三种方法,其他方法将来有机会咱们再逐个介绍。
地图展示的是空间差异。下一节,我们将聚焦在时间差异,看看疫情发展的时间序列。

